2016 年 12 月 16 日,清华大学生命学院、结构生物学高精尖创新中心施一公教授研究组于《科学》(Science)杂志就剪接体的结构与机理研究再发长文(Research Article),题为《酵母剪接体处于第二步催化激活状态下的结构》(Structure of a Yeast Step II Catalytically Activated Spliceosome),报道了酿酒酵母(Saccharomyces cerevisiae)剪接体在即将开始第二步剪接反应前的工作状态下的三维结构,阐明了剪接体在第一步剪接反应完成后通过构象变化起始第二步反应的激活机制,从而进一步揭示了前体信使 RNA 剪接反应(pre-mRNA splicing,以下简称 RNA 剪接)的分子机理。
由于真核生物中的基因编码区中存在不翻译成蛋白质的序列(称为内含子),染色体 DNA 转录出来的前体 mRNA(pre-mRNA)并不直接参与蛋白质翻译,而是需要先将其中的内含子片段去除,才能进入核糖体进行蛋白质合成。内含子的去除需要通过两步转酯反应来实现:首先,位于内含子序列中下游被称为分支点(branch point sequence)的序列中有一个高度保守的腺嘌呤核苷酸(A),其2’羟基亲核攻击内含子5’末端的鸟嘌呤(G),于是第一步反应发生,形成套索结构;然后,5’外显子末端暴露出的3’-OH 向内含子3’末端的鸟嘌呤发起攻击,第二步反应发生,两个外显子连在一起。通过这两步反应,前体信使 RNA 中数量、长度不等的内含子被剔除,剩下的外显子按照特异顺序连接起来从而形成成熟的信使 RNA(mRNA)(图1)。
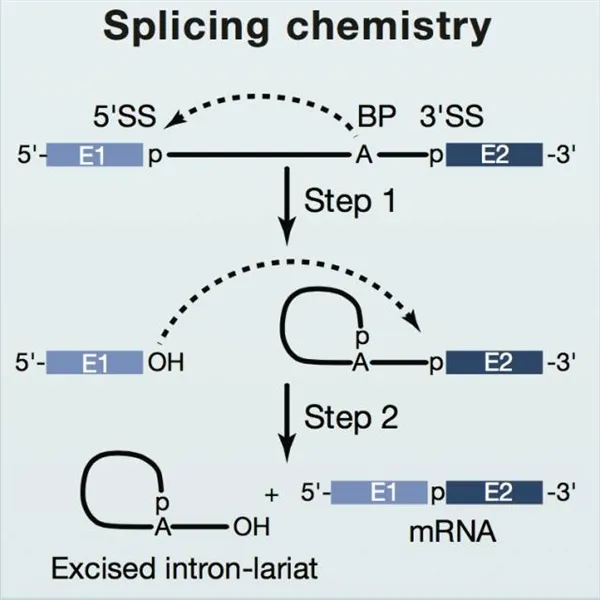
图 1 基因剪接反应示意图(图片来源:《Cell》)
这两步化学反应在细胞内是由一个庞大、复杂而动态的分子机器——剪接体催化完成的。对于每一个内含子来说,为了调控反应的各个基团在适当时机呈现合适的构象从而发挥其活性,剪接体各组分按照高度精确的顺序结合和解离,组装成一系列具有不同构象的分子机器,统称为剪接体。根据它们在 RNA 剪接过程中的生化性质,这些剪接体又被区分为B、Bact、B*、C、P、ILS 等若干状态。获取剪接体在组装、激活、催化反应过程中各个状态的结构是最基础也是最富挑战性的结构生物学难题之一。
2015 年 8 月,施一公研究组率先突破,在世界上首次报道了裂殖酵母剪接体处于 ILS 状态的 3.6 埃高分辨率结构。2016 年 7 月 22 日,施一公教授研究组在《科学》在线发表背靠背长文,首次报道了酿酒酵母剪接体分别处于激活状态(activated spliceosome,又称为 Bact complex)和第一步催化反应后(catalytic step I spliceosome,又称为 C complex)的近原子分辨率的剪接体结构,首次完整地展示了第一步转酯反应前后 pre-mRNA 和其中起催化作用的 snRNA 的反应状态,以及剪接体内部蛋白组分的组装情况。但是对于剪接体催化第二步转酯反应的细节,至今没有高分辨率的结构加以佐证。
在最新发表的《科学》长文中,施一公教授研究组捕获了性质良好的酿酒酵母剪接体样品,并利用先进的单颗粒冷冻电镜技术和高效的数据分类方法,重构出了总体分辨率分别为 4.0 埃的冷冻电镜结构,首次报道了酵母第二步催化激活状态下的剪接体结构。该结构的解析,进一步补充了 mRNA 剪接过程的关键信息,描述了从第一步转酯反应到第二步转酯反应过程中,剪接体催化反应活性中心内部组分的变化,以及关键蛋白的参与情况,为理解第二步反应所需的3’剪接位点是如何进入活性位点提供了重要的结构基础。值得关注的是,该结构的催化核心区域的分辨率达到 3.5 埃,第一次展示了转酯反应进行中的关键结构信息,填写了第二步转酯反应细节信息的空白。
2015 年 8 月至今,施一公教授研究组共报道了剪接反应中 5 个关键状态剪接体复合物的高分辨率结构,分别是 3.8 埃的预组装复合物 tri-snRNP、3.5 埃的激活状态复合物 Bact complex、3.4 埃的第一步催化反应后复合物 C complex、4.0 埃的第二步催化激活状态下的C* complex 以及 3.6 埃的内含子套索剪接体 ILS complex。这 5 个高分辨率结构所代表的剪接体状态,基本覆盖了整个剪接通路中关键的催化步骤,提供了迄今为止最为清晰的剪接体不同工作状态下的结构信息,大大推动了 RNA 剪接研究领域的发展。
施一公教授为本文的通讯作者;清华大学生命学院博士后结构生物学高精尖创新中心卓越学者闫创业、医学院四年级博士生万蕊雪以及生命学院二年级博士生白蕊为该文的共同第一作者;生命学院二年级博士黄高兴宇参与了这项研究;电镜数据采集于清华大学冷冻电镜平台,计算工作得到清华大学高性能计算平台、国家蛋白质设施实验技术中心(北京)以及荣之联董事长王东辉先生的支持。本工作获得了北京市结构生物学高精尖创新中心及国家自然科学基金委的经费支持。(来源:清华大学结构生物学高精尖创新中心)
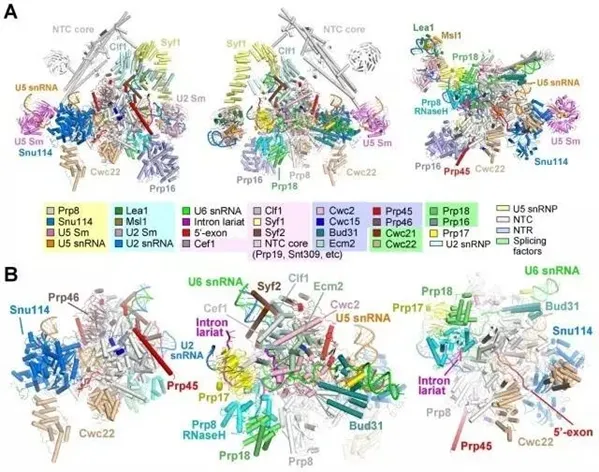
图 2 C* complex 三维结构示意图
本文来自清华大学结构生物学高精尖创新中心微信公众账号
